DNAからmRNAへ転写される反応のメカニズムを世界で初めて解明 -分子生物学のセントラルドグマ解明の手がかりをつかむ-
理化学研究所
Dmitry G. Vassylyev
横山 茂之
ウイルスからヒトに至る多くの生物は遺伝子;DNAを持っています。DNAは、主に、生命活動の維持に不可欠な、タンパク質を合成するための設計図として機能しています。DNA上の遺伝情報は、先ず、メッセンジャーRNA(mRNA)と呼ばれる物質へコピーされ、mRNAの情報をもとにタンパク質が作られます。この「DNA→mRNA→タンパク質」という細胞内における遺伝情報の流れは、生命の営みの基本的かつ普遍的な反応であるため、“分子生物学のセントラルドグマ”と呼ばれています。したがって、これらの反応の詳細なメカニズムを解明できれば、生命現象の根本を理解することができます。
DNAからmRNAへ転写される反応のメカニズムを世界で初めて解明
DNAからmRNAへコピーされるステップは「転写」と呼ばれ、遺伝子が働き始めるための最初の段階として重要です。RNAポリメラーゼは、転写反応を直接つかさどっている重要なタンパク質です。RNAポリメラーゼは、まず、プロモーターと呼ばれる、遺伝子の上流に位置する特徴的な部分に結合します。そして、DNA上を下流に向かってスライドしながら、DNAの片方の鎖を鋳型とし、これに対する相補的な塩基;リボヌクレオチド*を順番に一つずつ正確につなげ、mRNAを合成します。
細菌から高等生物に至る多くのRNAポリメラーゼは多数のポリペプチド鎖(マルチサブユニット*)から成る巨大で複雑な構造をしたタンパク質であるのに対し、細菌に感染するウイルスの一種である、T7ファージ由来のRNAポリメラーゼ(T7RNAポリメラーゼ;分子量98 kDa)は単一のポリペプチド鎖から成る、比較的小さな、単純な構造をしたタンパク質です。興味深いことに、両者共に同様のメカニズムで転写反応を行っていることが明らかになってきました。研究グループでは、T7RNAポリメラーゼをモデルとして用い、mRNAが合成されるメカニズムを世界で初めて解明しました。
mRNAが合成されるメカニズム
RNAポリメラーゼは、mRNAの構成単位の前駆物質であるリボヌクレオチド三リン酸を認識して加水分解し、RNA分子の末端につなげます。この反応が繰り返されて、mRNAが合成されます。研究グループは、リボヌクレオチドの一つであるATPの非加水分解型疑似物質;α,β-メチレンATP(AMPcPP)*を用い、mRNA合成の途上の状態の構造解析を試みました。先ず、転写反応を反映するようにデザインした18塩基対からなるDNA:RNAハイブリッド分子、AMPcPP、及び、T7RNAポリメラーゼから成る複合体の結晶を単離しました。そして、この複合体の結晶に対し、大型放射光施設SPring-8の理研構造ゲノムビームライン;BL26を用いてX線回折データを収集し、その結果を基に複合体の立体構造を決定しました。SPring-8の高輝度の放射光を用いることによって3.0Å(オングストローム)*(=3.0x10-10メートル)以上という、分解能の高い(精密な)X線回折データを得ることができました。複合体の立体構造を解析した結果、AMPcPPは、DNA:RNAハイブリッド分子上のDNA鎖の相補ヌクレオチドと塩基対を形成し、さらに、T7RNAポリメラーゼ分子上のYヘリックス*と呼ばれる部分に結合していました。興味深いことに、本複合体中のT7RNAポリメラーゼ分子は、非活性型である“開いた”構造(図1の左)をしていました。すなわち、T7RNAポリメラーゼは、活性型である“閉じた”構造(図1の右)へ変化する前に、Yヘリックス;“プレセレクションサイト”で正しいヌクレオチドを識別していると考えられます。一方、T7RNAポリメラーゼは、DNAの複製をつかさどる酵素の一つであるDNAポリメラーゼ?と構造上の類似点がありますが、DNAポリメラーゼ?の場合は、活性型である“閉じた”構造に変化した際に正しいヌクレオチド(この場合はDNAの構成単位であるデオキシリボヌクレオチド)が識別されるので、T7RNAポリメラーゼによるRNA合成のメカニズムと異なっています。
T7RNAポリメラーゼ上の“プレセレクションサイト”に相当すると考えられる部分がマルチサブユニット型RNAポリメラーゼ*にも見つかっているので、今回の発見は、生物種を超えた普遍的な転写反応の原子レベルでの理解につながると考えられます。
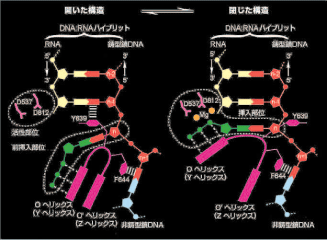
図1.リボヌクレオチド三リン酸(緑)は、“開いた”構造を持つ非活性型T7RNAポリメラーゼ
分子中のYへリックス部分(赤紫)に結合する(左図)。
その後、T7RNAポリメラーゼは、活性型の“閉じた”構造へと変化し、mRNAを合成する(右図)。
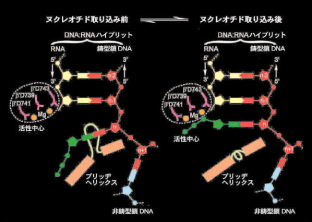
図2.マルチサブユニット型RNAポリメラーゼによるRNA合成のモデル。
リボヌクレオチド三リン酸(緑)は、先ず、ブリッヂへリックス部分に結合する(左図)。
ブリッヂへリックスの構造変化に伴い、mRNAが合成される(右図)。
今後の展開と応用
DNAからmRNAへの転写反応は、“分子生物学のセントラルドグマ”の最初のステップに相当します。今回、その基本的なメカニズムを原子レベルで明らかにしたことで、普遍的な生命現象の理解が一歩進んだといえます。現在、マルチサブユニット型RNAポリメラーゼを材料にして、さらに知見を深めるよう研究を進めています。
一方、RNAポリメラーゼはすべての生物の生命活動に必須なタンパク質であるため、抗生物質のターゲットになります。本研究で明らかになった立体構造から得られる知見を生かして、真核生物と原核生物のRNAポリメラーゼ構造の微妙な差異に着目し、病原性細菌を含む原核生物のポリメラーゼにだけ特異的に結合する化合物を作ることで抗生物質として利用できる可能性があります。このような新たな抗生物質や活性制御物質の創製といった医療への応用を目指した研究も飛躍的に進展するものと期待されます。
この成果は、米国の科学雑誌『Cell』の2月6日号に掲載されました。〈Temiakov D., et al. ,“Structural basis for substrate selection by T7 RNA polymerase”, Cell 116, 381-391 (2004).〉
本稿をまとめるにあたって、横山構造分子生物研究室 新海暁男先任研究員に大変お世話になりました。この場を借りて感謝いたします。
用語解説
●リボヌクレオチド
RNAの構成単位で、糖成分と塩基成分とから成る。RNAを構成する主な塩基成分は、アデニン(A)、グアニン(G)、シトシン(C)、ウラシル(U)の四種類である。RNAはこれらのリボヌクレオチドが一本鎖状に重合したポリヌクレオチドである。
●ATP
アデノシン5,-三リン酸の略で、RNAの構成単位の前駆物質の一つ。他の前駆物質として、GTP、CTP、UTPがある。
●オングストローム(Å)
長さの単位で、1オングストロームは1x10-10メートル(=0.1ナノメートル)。タンパク質の立体構造解析においては、解析した構造の分解能を表す単位として用いられ、数字が小さいほどより精度の高い高解像度の立体構造であることを示す。
●へリックス
タンパク質のとる高次構造の1つ。アミノ酸3.6個で1回転するらせん形構造で、アミノ酸1個あたり1.5のすすみをもつ。
●マルチサブユニット型RNAポリメラーゼ
原核細胞のRNAポリメラーゼの多くは、α,β,β,,σ,ωの5種類のサブユニットから成る、分子量約45万のタンパク質。真核細胞のRNAポリメラーゼの多くは、約10種類のサブユニットから成る、さらに複雑な構造を持つ。