生命に危機が迫ると機能する、高度好熱菌の新規転写因子を発見 -飢餓や酸化ストレスに対抗する遺伝子の発現を促進-(プレスリリース)
- 公開日
- 2008年08月28日
- BL26B2(理研 構造ゲノムII)
2008年8月28日
独立行政法人理化学研究所
本研究成果のポイント
○新規転写因子「SdrP」が14個の遺伝子発現を調節して、飢餓や酸化ストレスを回避
○生命を脅かす危機的状況を回避するメカニズムの一端を解明
○1つの細胞内のあらゆる生命現象をシステム全体として理解する一歩に
|
独立行政法人理化学研究所(野依良治理事長)は、85℃という高温で生育し、進化の起源に近いと考えられる高度好熱菌サーマス・サーモフィラス HB8株(T. thermophilus HB8)※1を「原子分解能で細胞全体の生命現象を理解するためのモデル生物」として利用し、新規転写因子「SdrP(Stationary-phase Dependent Regulatory Protein)」を発見しました。この転写因子は、増殖の定常期に発現量が増加し、飢餓や酸化ストレスなどに対抗するための14個の遺伝子の発現を制御していました。これは、理研放射光科学総合研究センター(石川哲也センター長)放射光システム生物学研究グループ(倉光成紀グループディレクター)の上利佳弘リサーチアソシエイト、新海暁男チームリーダーらが「高度好熱菌丸ごと一匹プロジェクト※2」で行った研究成果です。 (論文) |
1.背 景
研究グループでは、タンパク質をはじめとする生体分子の立体構造と機能に基づいて、1つの細胞におけるあらゆる生命現象を、システム全体として理解しようと研究を進めてきました。1999年には「高度好熱菌丸ごと一匹プロジェクト」を立ち上げ、モデル生物として、85℃という極限環境で生育できる高度好熱菌サーマス・サーモフィラス HB8株を選びました。その理由として、サーマス・サーモフィラス HB8株は好熱菌の中でも、(1)遺伝子数が約2,200個と少ない(ヒトは約23,000個、大腸菌は約4,500個)(2)厳しい環境に生きているためタンパク質が丈夫で、立体構造や機能を調べるのに都合がよい(3)遺伝子を操作する方法が確立されている、などのモデル生物に適した多くの特徴を持っているためです。
DNAが持つ生命の基本情報はRNAポリメラーゼによってmRNAへと転写され、その情報を基に、生体物質の合成や分解、栄養素の細胞内への取り込みや不要物質の細胞外への排出などの機能を持つさまざまなタンパク質が、リボソームで合成されます。細胞は、環境の変化や外部の刺激に応答する際に、転写因子と呼ばれるタンパク質を介して転写の調節を行っています。転写は、分子生物学のセントラルドグマ※5の最初のステップに相当するので、その詳細なメカニズムを解明すれば、あらゆる生物に共通する普遍的な生命現象の一端を理解できることになります。
モデル生物の1つとして長年研究されてきている大腸菌や枯草菌の転写因子は、全遺伝子数の1割弱を占めると考えられています。それに比べ、サーマス・サーモフィラスHB8株は、ゲノムサイズが約2.1 Mbp(1 Mbp : 100万塩基対)と小さい(ヒトは約3,000 Mpb、大腸菌は約4.6 Mbp)だけでなく、全遺伝子約2,200個に対する転写因子の数が約50個程度であるため、非常に少ない種類の転写因子からなる調節機構が働いていると考えられます。さらに、サーマス・サーモフィラスHB8株は、進化の起源に近いと考えられていることから、この菌株を用いた研究により、より基本的な転写調節機構が明らかになると期待できます。
2.研究手法と成果
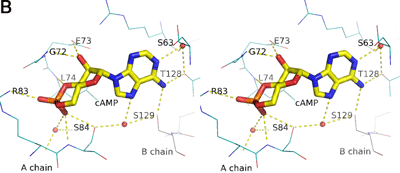
研究グループは、サーマス・サーモフィラス HB8株の機能未知転写因子の1つが定常期に発現量が特に増加することを発見し(図1)、このタンパク質をSdrP(Stationary-phase Dependent Regulatory Protein)と名付けました。SdrPは、多くの細菌がもつグローバル転写因子※6であるCRP/FNRファミリーに属します。このファミリーの転写因子の多くは、cAMP※7などの低分子補欠因子が結合するとDNAに結合できる活性型(DNA結合型)へと構造が変化します。
SdrPについて原子レベルの分解能でイメージングを行うため、大型放射光施設SPring-8の理研構造ゲノムII ビームラインBL26B2を用いてX線結晶構造を解析しました。その結果、SdrPは、cAMPなどの補欠因子が結合していないにもかかわらず、DNA結合型である大腸菌CRP-cAMP複合体と非常によく似た構造をしていました(図2)。また、SdrPは、多くのCRP/FNRファミリータンパク質と同様に、N末端ドメインとC末端ドメインから成り、C末端ドメインはDNA結合タンパク質特有の、2つのαヘリックスがβターンにより連結されている構造(ヘリックス-ターン-ヘリックス構造)をしていました。しかし、N末端ドメインのcAMP結合部位に相当する領域の構造を詳細に比較すると、SdrPでは、側鎖の大きいアミノ酸残基が存在しており、cAMPが結合できないことがわかりました(図3)。従って、SdrPは、cAMPなどの補欠因子と結合しなくともDNAに結合できる新規転写因子であると推測しました。
このSdrPを欠損させたsdrP遺伝子破壊株は、エネルギー源に乏しい最少培地での増殖が、野生株に比べて遅くなるという特徴を持っています(図4)。そこで、DNAマイクロアレイ※8を用いて、サーマス・サーモフィラスの野生株およびsdrP遺伝子破壊株における全遺伝子の発現解析を行い、SdrPの標的遺伝子を探索した結果、SdrPの働きによって転写が促進される14個の遺伝子を同定しました。これらの標的遺伝子あるいはオペロン※9が、実際にSdrPによって転写が促進されることを試験管内転写実験によって確認しました(図5)。その結果、立体構造から予想したとおり、SdrPは補欠因子なしで転写促進活性を示しました(図5)。また、野生株では、これらの遺伝子はいずれも定常期に発現が上昇していました(図6)。SdrPの標的遺伝子の多くは、酸化還元反応の調節や、エネルギー獲得といった生命活動の根源にかかわる遺伝子でした(表1)。
通常、細胞内は一定の還元的環境に保たれており、酸化ストレスによってタンパク質やDNAが損傷を受けると修復する機能を持ちます。一方、細胞は、電子伝達系に代表される一連の酸化還元反応や糖・アミノ酸の代謝により、エネルギーを獲得しています。しかし、定常期に入ると、エネルギーの枯渇、タンパク質やDNAの酸化傷害など、生命にとって危機的な状況になります。SdrPの発現が定常期に増加するのは、そのような危機的な状況から回避するための遺伝子群の発現を促進するためであると考えられます。
本研究により、生命を脅かす危機からの回避という、基本的生命現象の一端を明らかにすることができました。
3.今後の期待
研究グループでは、高度好熱菌サーマス・サーモフィラスHB8株を用いて、生物1個体におけるあらゆる生命現象をシステム全体として理解することを目指しています。そこで、サーマス・サーモフィラスHB8株でまだ機能が知られていない、推定約500種類の遺伝子の機能を分類するために、すでに作製済みの500種類以上の遺伝子破壊用のプラスミドを活用して、遺伝子破壊株の発現解析を進める計画です。同時に、野生株にさまざまな刺激を与え、その時に誘引される全mRNAの発現の変動をパターン解析することで、各種転写因子の標的遺伝子や機能を分子生物学的に明らかにしていきます。
研究グループはさらに、転写調節によって誘引される、細胞の形態や細胞内の金属イオンの分布など、細胞レベルでの変化を視覚的にとらえるために、原核細胞にも応用可能な、大型放射光施設SPring-8におけるサブミクロンオーダー(1μm以下の分解能)のイメージング技術を有効に利用したいと考えています。
このように遺伝子発現解析をはじめとする分子機能解析、原子分解能レベルのタンパク質のX線結晶構造解析と細胞内のイメージングを組み合わせることで、1つの細胞における基本的生命現象を“丸ごと一匹”でイメージできれば、ヒトを含めた高等動物における基本的生命現象の理解にもつながると考えています。
〈参考資料〉
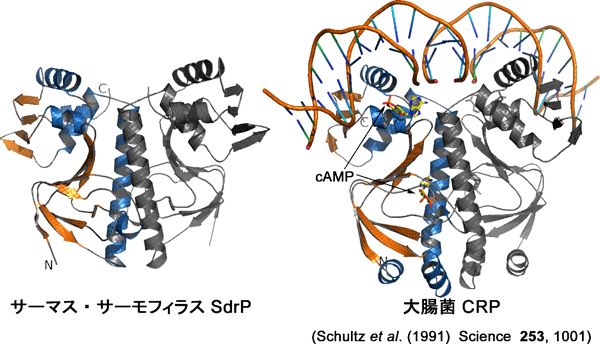 図2 サーマス・サーモフィラス SdrPと代表的なCRP/FNRファミリータンパク質 大腸菌 CRP(cAMPとの複合体)の立体構造
図2 サーマス・サーモフィラス SdrPと代表的なCRP/FNRファミリータンパク質 大腸菌 CRP(cAMPとの複合体)の立体構造CRP/FNRファミリーの転写因子の多くは、cAMPなどの低分子の補欠因子と結合することで、DNA結合型に構造変化すると考えられている。CRPファミリーの代表例である大腸菌 CRP(右)には、タンパク質1分子につき2つのcAMP結合サイト(プライマリ/セカンダリ)があり、このうちプライマリサイト(図下側)がDNA結合能を決定している。これまでに、cAMPが結合していないアポ型の構造は決定されていない。今回構造を決定したサーマス・サーモフィラス SdrP(左)は、補欠因子が結合していないにもかかわらず、DNA結合型である大腸菌 CRP-cAMP-DNA複合体と非常によく似た構造であった。
 |
|||||||
|
|||||||
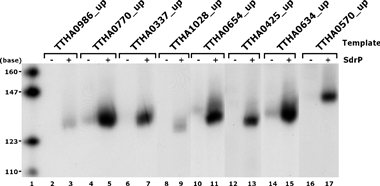 図5 標的遺伝子の試験管内転写実験
図5 標的遺伝子の試験管内転写実験DNAマイクロアレイを用いた遺伝子発現解析により、sdrP遺伝子を欠損させると発現量が有意に低下する遺伝子を複数発見した。そこでこれらの標的遺伝子候補について、RNAポリメラーゼによる試験管内転写実験を行い、SdrP依存的に転写が促進される14遺伝子(3つのオペロンを含む)(図6参照)を同定した。
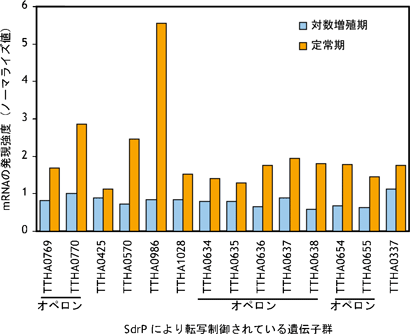 図6 各標的遺伝子の対数増殖期と定常期におけるmRNA発現量
図6 各標的遺伝子の対数増殖期と定常期におけるmRNA発現量サーマス・サーモフィラスHB8野生株の対数増殖期と定常期における全mRNAの発現量を、DNAマイクロアレイを用いて解析した結果、SdrPの標的遺伝子すべてのmRNAが定常期において増加していた。
遺伝子名 |
遺伝子の種類 |
解説・考察 |
|
1 |
TTHA0769 | タンパク質分解酵素 (アミノ酸配列から推測) |
細菌からヒトまで、幅広い生物種に存在する、トリプシン様のタンパク質分解酵素だと思われる。本酵素ファミリーのタンパク質は、栄養飢餓の条件では、アミノ酸を補給するためにタンパク質を分解しており、タンパク質の品質管理を行っている。 |
2 |
TTHA0770 | タンパク質分解酵素 (プロテアーゼ・ロン) |
細菌からヒトまで、幅広い生物種に存在している。細菌の場合、栄養飢餓の条件では、アミノ酸を補給するためにタンパク質を分解する。タンパク質の有効期限を調節しており、古くなり変性したタンパク質や、不要となったタンパク質を分解する。 |
3 |
TTHA0425 | 酸化還元酵素「NADH オキシダーゼ」 | 補酵素NADHをNAD+に酸化する。NADHの酸化還元反応は生体エネルギーの産生に関係している。 |
4 |
TTHA0570 | Glucose/Sorbosone 脱水素酵素(アミノ酸配列から推測) | NAD+を補酵素として糖を代謝し、エネルギーの産生に関与していると考えられる。TTHA0425タンパク質と共役している可能性がある。 |
5 |
TTHA0986 | 酸化還元酵素「チオレドキシン」 (アミノ酸配列から推測) |
チオレドキシンは、多くの生物種に存在し、S-S(ジスルフィド)結合を持つ種々の酵素の酸化還元を通して、その活性を調節する。定常期の細菌の細胞内ではDNAやタンパク質が酸化的ストレスにより損傷を受けることが知られているが、チオレドキシンは、生体内で発生した1重項酸素(1O2)やヒドロキシルラジカル(・OH)などの活性酸素種を消去する抗酸化物質として働く。 |
6 |
TTHA1028 | 酸化還元酵素 「ロダネーゼ」(立体構造から推測) |
ロダネーゼは、過酸化水素を還元して無毒化するチオレドキシンオキシダーゼ活性を持つ。 |
7 |
TTHA0634 | マグネシウムキラターゼ (アミノ酸配列から推測) |
細菌からヒトまで幅広い生物種に存在し、タンパク質にマグネシウムを配位させる酵素である。本遺伝子と同一オペロン上にあるTTHA0636遺伝子にコードされているタンパク質が金属を配位するモチーフを持つことから、TTHA0636にマグネシウムを配位する酵素であると推察できる。 |
8 |
TTHA0635 | ヌクレオチド転位酵素 (アミノ酸配列から推測) |
ヌクレオチドを付加する酵素である。本酵素群の1つであるポリA ポリメラーゼは定常期においてmRNAの3’末端にポリAを付加することで、mRNAの分解を促進する。 |
9 |
TTHA0636 | ヌクレオチド転位酵素 (立体構造から推測) |
TTHA0635タンパク質と複合体を形成すると考えられる。金属配位モチーフを持つ。 |
10 |
TTHA0637 | 未知 | 同一オペロン上の遺伝子にコードされているヌクレオチド転位酵素様タンパク質と関連した機能を持っていると推察できる。 |
11 |
TTHA0638 | 未知 | 同一オペロン上の遺伝子にコードされているヌクレオチド転位酵素様タンパク質と関連した機能を持っていると推察できる。 |
12 |
TTHA0654 | 未知 | 鉄-硫黄クラスターの代謝に関与している可能性のある領域を持つ。活性中心に鉄-硫黄クラスターを持つタンパク質には、酸化還元反応に関与しているものもある。 |
13 |
TTHA0655 | 転写因子 (アミノ酸配列から推測) |
現在、本転写因子の標的遺伝子を探索中である。 |
14 |
TTHA0337 | 未知 | 本研究結果から、細胞の危機的状況から回避するために機能している新規タンパク質と推察できる。 |
〈用語解説〉
※1 高度好熱菌サーマス・サーモフィラス HB8株
静岡県伊豆半島にある峰温泉から発見された、85℃という極限環境で生育できる細菌(バクテリア)。熱水中で生きている細菌(好熱菌)は全生物の共通祖先に近い位置にあり、原始生命の基本的特徴が凝縮されているといわれている。好熱菌1匹に起こる生命現象を理解することは、ヒトを含めたあらゆる生物の基本を解明し、「生命とは何か」という本質的な疑問を問い直すことにつながるとも考えられている。
※2 高度好熱菌丸ごと一匹プロジェクト
高度好熱菌サーマス・サーモフィラスHB8株を地球上のあらゆる生物の代表(モデル生物)とし、DNA、タンパク質、糖質、脂質、そのほか低分子の構造と機能に基づいて、1つの細胞におけるあらゆる生命現象をシステム全体として理解する学問基盤の構築を目指すプロジェクト。このプロジェクトは以下の4段階で進行すると想定しており、SPring-8においてはイメージングに関連した研究を行う。
第1段階:タンパク質など細胞を構成する分子の、1つの細胞全体の立体構造解析
第2段階:タンパク質など細胞を構成する分子の、1つの細胞全体の機能解析
第3段階:細胞内のそれぞれのシステム(複数分子のネットワーク関係)の解析
第4段階:細胞全体のシミュレーション
※3 大型放射光施設SPring-8(スプリングエイト)
理研が所有する、兵庫県の播磨科学公園都市にある世界最高の大型放射光施設。SPring-8の名前はSuper Photon ring-8GeVに由来する。放射光(シンクロトロン放射)とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げたときに発生する、細く強力な電磁波のことである。SPring-8では、遠赤外から可視光線、軟X線を経て硬X線に至る幅広い波長域で放射光を得ることができるため、原子核の研究からナノテクノロジー、バイオテクノロジー、産業利用や科学捜査まで幅広い研究が行われている。SPring-8は日本の先端科学・技術を支える高度先端科学施設として、日本国内外の大学・研究所・企業から年間1万4,000人以上の研究者が利用している。
※4 サブミクロンオーダーの細胞イメージング技術
細菌などの原核生物にも利用可能なイメージング技術。真核細胞は10~100μm前後の大きさをもつため、波長数百nmの光を利用する蛍光法が利用可能である。しかし細胞の大きさが数μmしかない細菌では、蛍光法では非常に粗いモザイク状にしか可視化できないため、蛍光法のイメージングはあまり行われていない。そのため、波長がはるかに短いX線を利用したイメージングの技術を細菌に応用することを試みている。
※5 セントラルドグマ
ワトソンと共にDNAの2重らせん構造を発見したクリックにより提唱された生命の基本的な原則。遺伝情報が記憶されているDNAがRNAへ転写された後、細胞小器官であるリボソームにて翻訳されてタンパク質が合成される一連の流れのこと。
※6 グローバル転写因子
数多くの遺伝子の発現を調節している転写因子。代表的なものとして、cyclic AMP receptor protein (CRP)/ fumarate and nitrate reduction regulator (FNR)ファミリーが知られる。
※7 cAMP
細胞内でのエネルギー伝達体であるATPから合成される。種々の外界情報を細胞内の標的分子に伝える細胞内シグナル伝達のセカンドメッセンジャーとして働き、多くのタンパク質リン酸化酵素を活性化するほか、糖や脂質の分解などの多くの生物学的過程をコントロールする。
※8 DNAマイクロアレイ
スライドガラスやシリコンの基盤上に、数万から数十万個の遺伝子の断片を固定し、それらの遺伝子断片と、調べたい細胞から調製したmRNAの相補DNA(cDNA)との結合を解析することによって、遺伝子の発現を調べる方法。数万から数十万個の遺伝子の発現を同時に調べることが可能であるため、細胞内のすべての遺伝子の発現を調べるなど、網羅的解析を行う際に威力を発揮する。
※9 オペロン
1つのプロモーターの制御下に複数の遺伝子が遺伝子群として連なり、1つの転写単位を形成しているもの。
|
(問い合わせ先) 播磨研究推進部 企画課 (報道担当) (SPring-8に関すること) |
- 現在の記事
- 生命に危機が迫ると機能する、高度好熱菌の新規転写因子を発見 -飢餓や酸化ストレスに対抗する遺伝子の発現を促進-(プレスリリース)
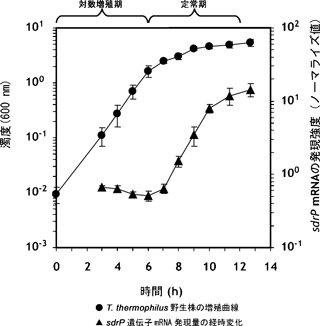 図1 サーマス・サーモフィラス HB8株の増殖とsdrP mRNA発現量の経時変化
図1 サーマス・サーモフィラス HB8株の増殖とsdrP mRNA発現量の経時変化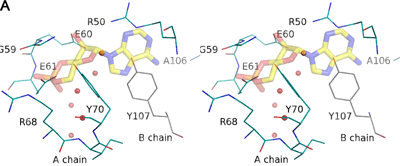
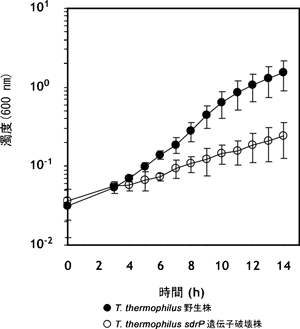 図4 最少培地中におけるサーマス・サーモフィラス HB8野生株とsdrP遺伝子破壊株の増殖曲線
図4 最少培地中におけるサーマス・サーモフィラス HB8野生株とsdrP遺伝子破壊株の増殖曲線