タンパク質生合成の最初の瞬間を捉えることに成功 - リボソームとシャイン・ダルガーノ(SD)配列の超分子複合体 -(プレスリリース)
- 公開日
- 2007年03月15日
- BL41XU(構造生物学I)
- BL44XU(生体超分子複合体構造解析)
平成19年3月15日
独立行政法人理化学研究所
マックスプランク研究所(ドイツ)
本研究成果のポイント
● リボソームがSD配列を認識する機構をX線結晶構造解析により分子レベルで解明
● mRNAはSD配列を介してリボソームに結合した後に3’側(後方)へと移動
● タンパク質生合成の開始制御にmRNAの2状態の平衡が重要な役割を果たす
|
独立行政法人理化学研究所(野依良治理事長)とドイツのマックスプランク研究所(ペーター・グルース会長:President Dr. Peter Gruss)は共同で、タンパク質生合成の鋳型となるメッセンジャーRNA(mRNA)が持つ「シャイン・ダルガーノ(SD)配列」が、原核生物※1リボソーム※2の30Sサブユニット※3と超分子複合体を形成した状態を捉えることに成功し、これまで知られていなかった結合部位と結合様式を解明しました。これは理研ゲノム科学総合研究センター(榊佳之センター長)タンパク質構造・機能研究グループの横山茂之プロジェクトディレクター、竹本千重上級研究員、上西達也リサーチアソシエイトとマックスプランク研究所のパオラ・フッチーニ(Dr. Paola Fucini)のグループによる研究成果です。 (論文) |
1.背 景
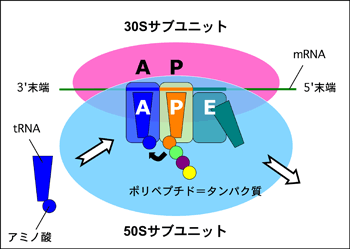
細胞内の生命現象の主役であるタンパク質は、あらゆる生物で共通の細胞内小器官「リボソーム」で作られています。リボソームは、DNAからメッセンジャーRNA(mRNA)に転写された遺伝情報を、トランスファーRNA(tRNA)を用いて解読し、対応するアミノ酸を連結することで、タンパク質を合成します(図1)。リボソームは、4,500残基にも及ぶRNA分子と50種類以上のタンパク質が複雑に組み合った超分子複合体であり、サブユニットと呼ばれる大小2つの塊に分けることができます。このうち小(30S)サブユニットは、約1,500残基のRNA分子と20種類以上のタンパク質で構成され、タンパク質生合成の鋳型となるmRNAが通るための溝を持っています。このmRNAの遺伝情報をタンパク質に正しく翻訳するためには、まず読み始めの位置を正しく決定することが重要です。すなわち、タンパク質生合成の開始段階において、mRNAの開始コドンが開始用のtRNA のアンチコドン(34番目から36番目の3文字の塩基配列)と正しく対合する必要があります。その最初のステップとして、mRNAが30Sサブユニットに結合する際に重要な役割を果たすのが、「シャイン・ダルガーノ(SD)配列」です。
1974年にオーストラリアのJ・シャインとL・ダルガーノは、バクテリアに感染するバクテリオファージのmRNAが、開始コドンの上流にプリンに富んだ配列を共通に持つことを見出しました。さらに、30Sサブユニットの主成分である16S リボソームRNA(rRNA)が、3’末端に相補的な配列を持つことから、両者が塩基対を形成して結合することにより、mRNAを鋳型としたタンパク質の生合成が促進される、という仮説を提案しました。その後、この仮説は多くの科学者によって証明され、これらの配列は発見者の名前から、mRNA側がシャイン・ダルガーノ(SD)配列、16S rRNA側がアンチ・シャイン・ダルガーノ(anti-SD)配列と呼ばれています(図2)。しかし、実際にどこでどのようにSD配列がanti-SD配列と相互作用しているのかは不明でした。
2.研究手法と成果
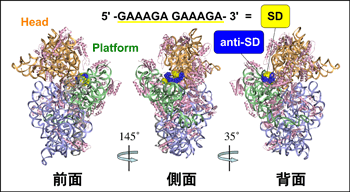
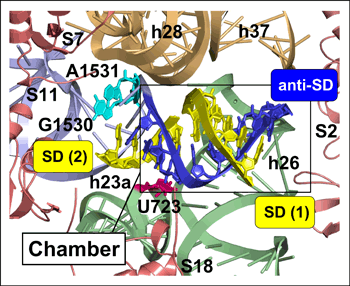
研究グループは、SD配列がリボソームの30Sサブユニットに認識されている瞬間を捉えるために、プリンに富む6残基のオリゴヌクレオチド(5’-GAAAGA-3’)が30Sサブユニットに結合している超分子複合体のX線結晶構造解析を行いました。具体的には、高度好熱菌Thermus thermophilus HB8(サーマス サーモフィラス エイチビーエイト)の30Sサブユニットを精製し、オリゴヌクレオチド存在下で得られた結晶を用いて、放射光科学研究施設Photon Factory(大学共同利用機関法人高エネルギー加速器研究機構)のBL-5AとNW12A、大型放射光施設(SPring-8)の構造生物学IビームラインBL41XUと生体超分子複合体構造解析ビームラインBL44XU、スイスのSwiss Light SourceのX06SAの各ビームラインでX線回折実験を行い、3.3Å(オングストローム)の分解能で立体構造を決定しました(図3)。まず明らかになったのは、30Sサブユニットのheadとplatformと呼ばれるドメイン(大きな構造単位)の間に、2重らせん構造がすっぽりとはまり込んでいることでした。詳細に調べてみると、2分子のオリゴヌクレオチドが縦列してSD配列を模し、anti-SD配列と塩基対を形成していることがわかりました。この2重らせん構造(SDへリックス)は、研究グループが「chamber」と名付けた空間に収まっており、その認識には16S rRNAの高度に保存されたヌクレオチド残基や2重らせん構造が関与していました(図4)。
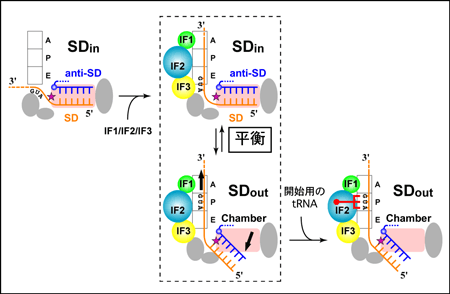
一方、この結果から予測されるmRNAの開始コドンの位置は、そのままでは開始用のtRNAのアンチコドンとの相互作用には適さないものでした。しかし、mRNA の30Sサブユニットへの結合についてこれまでに報告されている実験の結果を考慮すると、「mRNAはSD配列を介して30Sサブユニットに結合した後、開始コドンを含む下流の領域が30Sサブユニットの溝に結合するのに伴って、3’側(後方)に移動する」と解釈できることがわかりました。またこの際には、chamber内(in)に収まったSDへリックスがその外(out)に出る必要があります。そこで研究グループは、mRNAがこれらの2状態(SDinおよびSDout)の平衡で存在しており、タンパク質生合成の開始制御に重要な役割を果たしていることを、初めて提唱しました(図5)。
3.今後の展開
タンパク質生合成を正確に行うためには、その開始段階は非常に重要です。もしmRNAの遺伝情報を読み始める位置がずれてしまうと、異常なタンパク質が蓄積し、細胞にとって毒となる可能性もあります。逆に、SD配列を介したタンパク質生合成の開始機構は、多くの病原菌が属する原核生物に特有のものなので、それを特異的に阻害する物質をSBDD※8などにより探索することができれば、真核生物であるヒトには無毒な抗生物質としての応用も考えられます。その点でも、研究グループがSDへリックスの結合部位および結合様式を高分解能で決定したことや、タンパク質生合成の開始制御におけるSDinとSDoutの平衡の重要性を提唱したことは、非常に大きな意味を持ちます。
今後、長いmRNAやタンパク質生合成の開始段階に関与する因子との相互作用が、リボソームに結合した超分子複合体のX線結晶構造解析などにより進み、研究グループが提唱した仮説が証明されれば、既存の分子生物学の教科書を書き換えるような成果につながることが期待されます。
<参考資料>
<用語解説>
※1 原核生物
生物の分類のひとつであり、細胞に核を持たない原核細胞からなる生物を指す。
※2 リボソーム
細胞内に存在するタンパク質合成を担う超分子複合体で、リボソームRNAとリボソームタンパク質からなる。
※3 30Sサブユニット
原核細胞のリボソームは、沈降係数(大きさの指標)から50Sおよび30Sと呼ばれる大小2つのサブユニットから構成されている。
※4 バクテリオファージ
ウイルスのうち、細菌に感染するもの。
※5 開始コドン
mRNAのコドン(3文字の塩基配列)のうち、タンパク質合成の開始を指定するもので、メチオニンに対応するAUGが使われることが多い。
※6 プリン
DNAやRNAを構成するヌクレオチド(残基)のうち、アデニンやグアニンの塩基を総じてプリンと呼ぶ。
※7 高分解能
Å[オングストローム:1×10-10メートル(=0.1ナノメートル)]の単位を用いて表し、この数字が小さいほど分解能が高く、より精度の高い高解像度であることを示す。
※8 SBDD
Structure Based Drug Design(立体構造情報に基づく薬剤設計)の略。ある疾病関連タンパク質が特定された場合、その立体構造から引き出される情報を利用することにより、合理的な薬剤設計を行うことを可能にする。PCクラスター等の大規模計算機システムとの組み合わせにより、短期間・低コストでの創薬が可能になる。
|
<本件に関する問い合わせ先> 独立行政法人理化学研究所 横浜研究所 タンパク質構造・機能研究グループ 横浜研究所推進部 企画課 (報道担当) <SPring-8についての問い合わせ先> 財団法人高輝度光科学研究センター 広報室 |
- 現在の記事
- タンパク質生合成の最初の瞬間を捉えることに成功 - リボソームとシャイン・ダルガーノ(SD)配列の超分子複合体 -(プレスリリース)