自然免疫応答における細胞質センサーNOD2受容体の構造解明(トピック)
- 公開日
- 2016年06月13日
- BL32XU(理研 ターゲットタンパク)
- BL41XU(構造生物学I)
平成28年6月10日
東京大学
研究のポイント
• 細胞内に発現し、細菌の細胞壁の構成成分であるムラミルジペプチド注1を認識し、自然免疫を活性化させるNOD様受容体2(NOD2)と呼ばれるタンパク質の立体構造を解明しました。
• LRRドメインにリガンド認識に関わると推測されるポケットが存在しました。
• NOD2に数多く報告されるSNPs注2と疾患との関連について考察しました。
|
東京大学大学院薬学系研究科の清水敏之教授、大戸梅治講師、前川早貴子大学院生、同医科学研究所の三宅健介教授、柴田琢磨助教らの研究グループは、細菌の細胞壁の構成成分であるムラミルジペプチドを認識して自然免疫応答を活性化するNOD2受容体の詳細な立体構造を世界で初めて明らかにしました。 雑誌名:Nature Communications |
研究の背景
細菌やウイルスなどの病原体の侵入に対して即座に反応する感染防御機構として、私たちの体には自然免疫機構が備わっています。NOD様受容体(NLR)は細胞内に発現する自然免疫受容体ファミリーであり、自然免疫を制御する役割を担っています。一部のNLRはインフラマソームと呼ばれるタンパク質複合体を形成することで、活性化体として機能します。ヒトには22種類のNLRが存在しています。
NLRの一員であるNOD2は細菌の細胞壁の構成成分であるムラミルジペプチドを認識し活性化型の多量体を形成すると考えられています。最終的に、転写因子NF-κBを活性化することで、自然免疫応答を引き起こします。NOD2には多くの一塩基多型(SNPs)が報告されており、ブラウ症候群やクローン病といった自己炎症性疾患に関連しています。しかし、これまでNOD2の構造情報はなく、リガンド認識機構や、活性化機構、変異と疾患との関連についての詳細は不明でした。
研究の方法と結果
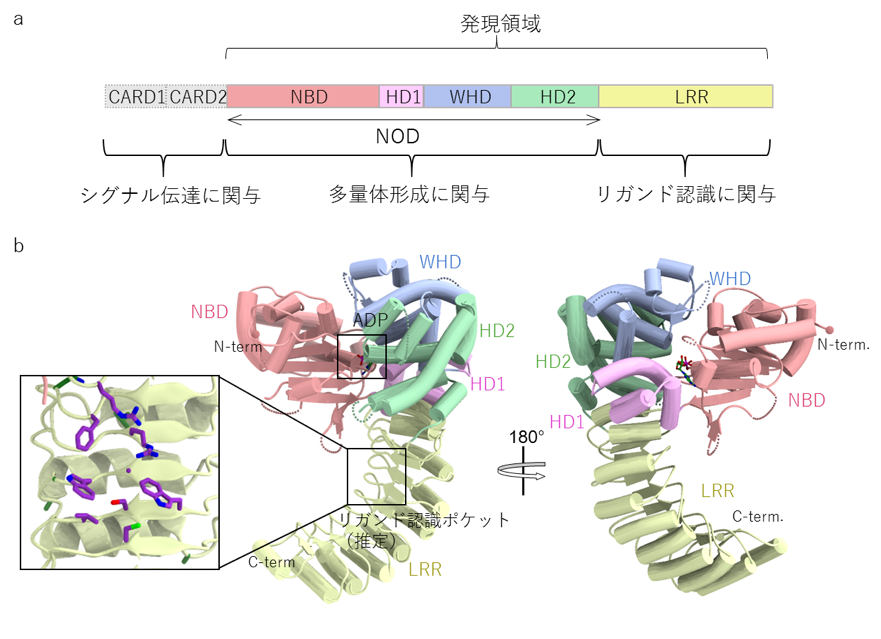
本研究グループは、シグナル伝達に関わるN末端側のドメイン(CARD)を欠損させたNOD2 (ラビット由来)(図1a)を大量に調製することに成功しました。得られたタンパク質を結晶化し、NOD2とADPとの複合体の結晶構造を明らかにしました。結晶からの回折データの取得には、大型放射光施設SPring-8のBL41XU、及びBL32XUの強力なX線を使用し、2.3 Å(オングストローム, 1 Åは0.1ナノメートル)という高い分解能での構造決定を実現しました。
構造解析の結果、ADPはNOD2のドメイン間の相互作用を媒介するように結合しており(図1b)、類似タンパク質との構造比較からNOD2は不活性化型のコンフォメーションであると結論づけました。また、リガンド認識に関連するとされているLRRドメインの凹面にポケットが存在していました(図1b左)。このポケットの構成残基の変異体実験から、このポケットがムラミルジペプチドの認識に関与していることが示唆されました。
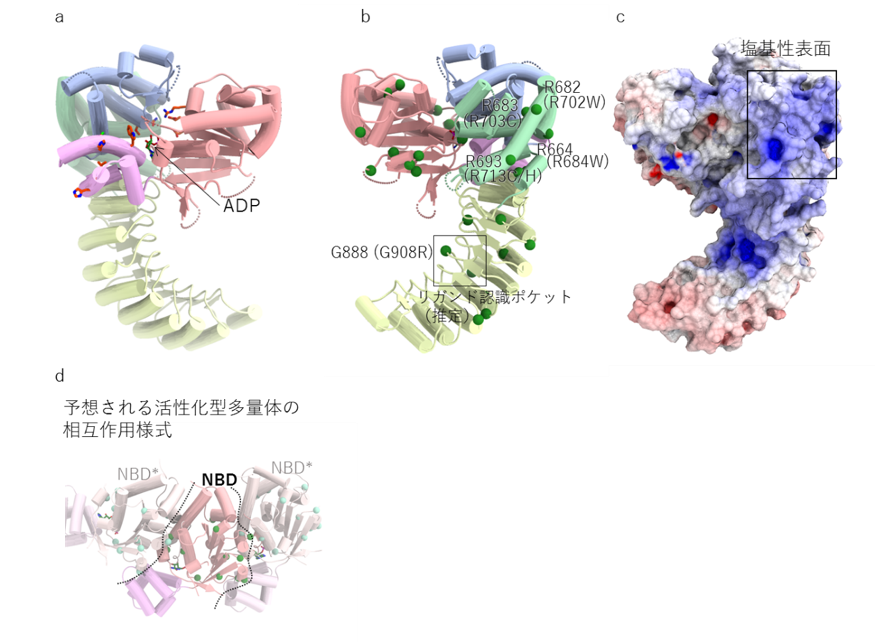
NOD2の恒常活性化型の変異がブラウ症候群や若年性サルコイドーシスの原因となることが確認されています。これらの変異残基は不活性化型NOD2のドメイン間の相互作用界面に集中しており(図2a)、変異導入で不活性化型NOD2の構造を不安定化し、NOD2の恒常活性化を引き起こすともの考えられます。
一方で、クローン病や潰瘍性大腸炎に関連があると報告される変異の一部はNOD2の機能欠損型の変異体であることが分かっており、これらの変異残基はNOD2全体に分布していました(図2b)。主に、次の3箇所に分類することができ、下記に示すような機構でNOD2の活性化を阻害しているものと推測できます。
1)塩基性表面を形成するような箇所(図2b, c)。NOD2は膜との相互作用が活性化に関係しているという報告があり、膜との相互作用の阻害により、NOD2の活性化が阻害されると考えられます。
2)NOD2が活性化型複合体を形成する際の、相互作用界面になると考えられる箇所(図2d)。変異導入で活性化型複合体の形成を阻害すると考えられます。
3)推定されるリガンド認識ポケットの近傍(図2b)。クローン病に関連する変異の中でも報告例の多いG908R(ヒトの残基番号)の残基は位置しており、大きなアルギニン残基に置換されることで、ポケットの構造が変化し、リガンド認識が阻害されるのではないかと考えられます。
研究の意義
本研究では、これまで未知であったNOD2の不活性化型構造を決定し、リガンドが結合すると推測される部位や、NOD2に数多く報告されているSNPsと疾患との関連を考察することができました。NOD2の構造情報は、NOD2のより詳細な活性化機構や、リガンド認識機構を解明していく上で重要な構造基盤となると期待されます。
a)NOD2のドメイン構成
略称は下記の通りになっている
CARD: Caspase recruitment domain
NOD: Nucleotide-binding and oligomerization domain
NBD: Nucleotide binding domain HD1: Helical domain 1 WHD: Winged helix domain
HD2: Helical domain 2 LRR: Leucine-rich repeat
発現領域と、各ドメインの機能を図中に示した。
b)NOD2の全体構造
リガンド認識ポケット(推定)周辺の拡大図を左に示し、ポケット構成残基を紫のスティックで表示している。
a)NOD2の全体構造にブラウ症候群と若年性サルコイドーシスの原因変異をオレンジのスティックでマッピングしている。ADPはスティックで表記している。ドメインの色は図1のものに従う。
b)NOD2の全体構造にクローン病や潰瘍性大腸炎に関連する変異を緑の球でマッピングしている。塩基性の表面を形成する残基と、リガンド認識ポケット近傍のG888がラベルしてある。括弧内はヒトの残基番号を表す。
c)bと同様の方向から見たNOD2の静電ポテンシャル図。青が正電荷、赤が負電荷を表しており、塩基性表面を形成していることがわかる。
d)NOD2と類似タンパク質であるCED4注4の構造に基づいて、NOD2の活性化型多量体におけるNODドメインの相互作用を予測した。中央のNOD2分子のNBDとHD1ドメインをそれぞれピンクと紫で示し、隣接するNOD2分子のNBDドメインを薄いピンクと紫で示した。破線は相互作用界面を表している。クローン病や潰瘍性大腸炎に関連する変異を緑の球でマッピングしてあり、相互作用界面に分布していることがわかる。
【用語解説】
注1. ムラミルジペプチド:
Muramyl dipeptide。グラム陽性菌、陰性菌両方の細胞壁の構成成分。N-acetylmuramic acidとL-alanine D-isoglutamine ジペプチドが結合した構造をとる。MDPとも呼ばれる。
注2. SNPs :
Single Nucleotide Polymorphism、一塩基多型。ある生物種集団のゲノム塩基配列中に一塩基が変異した多様性が見られること。しばしば、疾患のリスクファクターや原因になる。
注3. NF-κB:
阻害タンパク質と会合した不活性化型で細胞質に存在しているが、シグナルを受けると、阻害タンパク質が分解され、活性化される。活性化したNF-κBは核内に移行し、サイトカインなどの発現を誘導し炎症応答や細胞死を引き起こす。
注4. CED4:
Caenorhabditis elegans death protein。線虫由来のアポトーシスを開始させるために必要なタンパク質。
|
【問い合わせ先】 (SPring-8に関すること) |
- 現在の記事
- 自然免疫応答における細胞質センサーNOD2受容体の構造解明(トピック)
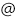 mol.f.u-tokyo.acjp
mol.f.u-tokyo.acjp